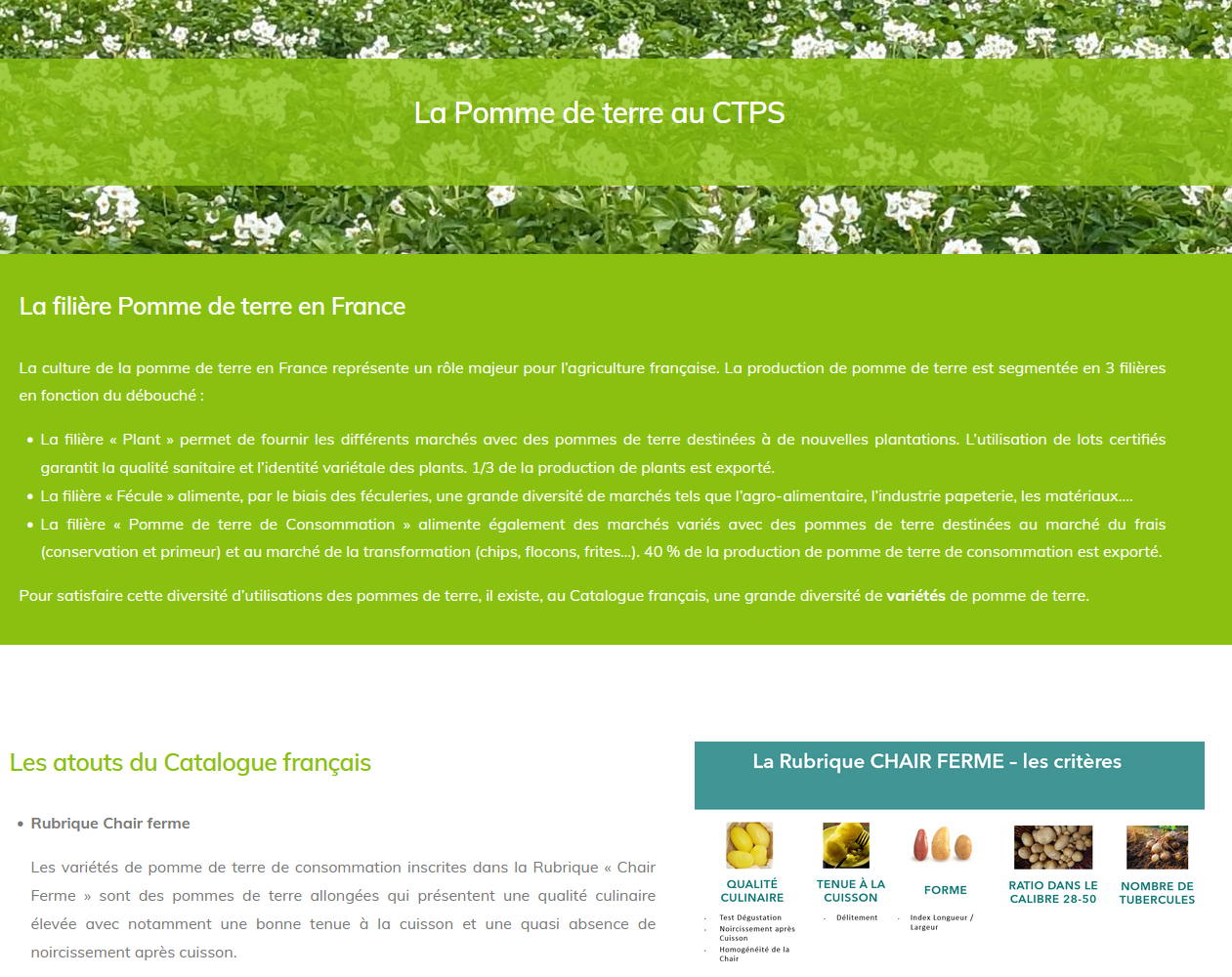
Un nouveau test RT-qPCR pour l’identification des virus de la jaunisse de la betterave
La jaunisse de la betterave sucrière est un complexe de quatre virus appartenant à trois genres viraux qui peuvent être présents en mono-infection ou en co-infection :
- BYV (Beet Yellows Virus), appartenant aux Clostérovirus.
- BChV (Beet Chlorosis Virus) et BMYV (Beet Mild Yellowing Virus), appartenant aux Polérovirus.
- BtMV (Beet Mosaic Virus), appartenant aux Potyvirus.

Cette maladie est principalement transmise par le puceron vert du pêcher (Myzus percisae) et entraîne des pertes de rendement pouvant aller jusqu’à 50%. Depuis 1993, les populations de pucerons étaient contrôlées par l’utilisation de néonicotinoïdes (NNI), cependant leur utilisation est interdite depuis 2018.
Le GEVES coordonne un projet collaboratif du Plan National de Recherche et Innovation (PNRI) : Yellows Resisbeet, avec deux objectifs principaux :
- Mettre au point un protocole d’évaluation des résistances/tolérances variétales de la betterave sucrière aux quatre virus responsables de la jaunisse.
- Définir un ou des modèles statistiques permettant d’estimer la productivité des variétés dans des essais en présence de jaunisses.
Dans le cadre de ce projet, il est important d’avoir un test de détection efficace pour les quatre virus. En effet, la méthode ELISA ne permet pas de distinguer les deux Polérovirus (BChV et BMYV). Pour répondre à cette problématique, le pôle Détection du BioGEVES, en collaboration avec l’INRAE de Colmar (équipe de Véronique Brault – projet PNRI ProViBe), a donc développé une méthode de détection et d’identification des quatre virus responsables de la jaunisse de la betterave par RT-qPCR multiplexe.
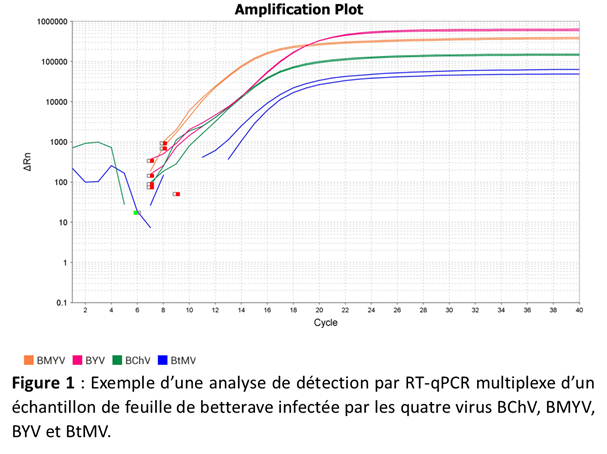
Quatre paires d’amorces et quatre sondes TaqMan ont été conçues pour cibler respectivement et spécifiquement les quatre virus BChV, BMYV, BYV et BtMV. Les quatre sondes TaqMan ont été chacune marquées par un fluorophore différent, ce qui a permis de développer une méthode RT-qPCR multiplexe donnant la possibilité de détecter et d’identifier les quatre virus en une seule réaction RT-qPCR (Figure 1).
La méthode est actuellement en fin de validation, et des critères de performance tels que la spécificité analytique, correspondant à la capacité d’une méthode à détecter le bioagresseur cible (inclusivité) en excluant les bioagresseurs non-cibles (exclusivité), et la sensibilité analytique, correspondant à la plus petite quantité de bioagresseur cible pouvant être détectée, ont été évalués.
Ainsi, il a été démontré que les taux d’inclusivité et d’exclusivité sont de 100% pour toutes les amorces et les sondes, à l’exception du taux d’exclusivité des amorces et de la sonde ciblant BYV qui est de 83%. La sensibilité de la méthode RT-qPCR multiplexe a été comparée avec celle de la méthode actuellement utilisée au GEVES, l’ELISA, et il a été démontré que la RT-qPCR multiplexe est 100 à 10 000 fois plus sensible que l’ELISA en fonction du virus.
Par ailleurs, cette méthode d’identification par RT-qPCR multiplexe a également été partagée avec deux laboratoires de recherche belges intéressés par cet outil dans le cadre de leurs projets respectifs :
- Le Centre wallon de Recherches agronomiques (CRA-W), dans le cadre du projet Virobett qui a pour objectif l’étude des épidémies des différents virus de la betterave et le développement d’une gestion de la jaunisse de la betterave.
- Gembloux Agro Bio-Tech Université de Liège, dans le cadre d’un projet portant aussi sur la jaunisse de la betterave afin d’adapter la méthode à la détection et l’identification des quatre virus directement sur pucerons.
Une fois que la méthode sera définitivement validée, elle sera proposée à la prestation au GEVES, des publications dans des revues scientifiques sont également envisagées en collaboration avec nos partenaires.