Mise au point d’une méthodologie qui utilise le marquage SNP pour optimiser la gestion des collections de référence de colza d’hiver dans le cadre des études DHS
Le colza (Brassica napus) est la deuxième plante oléagineuse la plus produite dans le monde et sa culture est en constante augmentation. Reflet de l’importance économique croissante de l’espèce, le nombre de variétés inscrites au catalogue a augmenté de manière significative ces dix dernières années, en majorité des hybrides, entraînant une augmentation substantielle de la taille de la collection de référence. Chez le colza, l’expression des caractères phénotypiques est très sensible aux variations environnementales. Pour tenir compte de cette forte variabilité interannuelle lors des examens DHS, l’ensemble de la collection de référence doit être redécrit chaque année. Cela représente d’énormes défis techniques et logistiques en termes de planification et d’infrastructure et des coûts importants. Tous les offices d’examen en Europe qui effectuent des tests DHS sur le colza d’hiver rencontrent des difficultés en raison de la taille grandissante des essais au champ et de la surcharge de travail que cela entraîne. Le GEVES (France) et le BSA (Allemagne) ont mené un projet commun avec pour objectif de développer de nouvelles approches qui combinent informations génétiques et phénotypiques pour éviter d’avoir à redécrire l’ensemble de la collection de référence chaque année sans compromettre la qualité des essais DHS. Le nouveau modèle devait être également compatible avec les différents systèmes utilisés en DHS pour gérer les collections de référence (GAIA ou COY-D).
Les objectifs du projet étaient donc :
– de génotyper ~80% de la collection de référence et de produire un jeu de données moléculaires cohérent pour environ 2000 variétés de colza d’hiver ;
– d’utiliser ces données moléculaires pour cribler et optimiser un jeu de 500 marqueurs SNP ;
– de concevoir et d’évaluer de nouvelles approches qui combinent informations génétiques et phénotypiques basées sur les marqueurs SNP et les données de terrain historiques accumulées au cours des 10-15 dernières années pour optimiser les essais DHS.
Ce projet nous a permis d’identifier un jeu optimisé de 360 SNP avec une bonne couverture génomique et un fort pouvoir discriminant. À partir de ces données, une nouvelle approche a été développée qui utilise l’analyse en réseau pour définir une « collection de référence optimale » (Figure 1). L’approche proposée s’appuie sur la théorie des graphes pour détecter des groupes de variétés apparentées sur la base de leur proximité au sein d’un réseau décrivant les relations génétiques des variétés entre elles. Dans ce modèle, des algorithmes sont utilisés pour identifier des groupes de variétés au sein du réseau en fonction de leur proximité (similarité génétique) ; seules les variétés de référence dans les groupes qui comprennent également des variétés candidates seront incluses dans les essais (Figure 1). En fonction de la méthode utilisée pour gérer habituellement les collections de référence (COY-D ou GAIA), le modèle pourrait permettre de réduire de 20 à 45 % la taille des essais en première année d’étude, sur la base des seuils préliminaires de distance génétique utilisés pour tester la méthode. Une vue schématique de la mise en œuvre du modèle est décrite à la Figure 2.
Cette méthode d’optimisation basée sur l’approche en réseau est implémentée dans R, un outil statistique open source et multiplateforme. Jusqu’à présent, la méthode n’a été testée qu’in silico, avec des seuils génétiques définis sur la base des collections franco-allemandes. Le jeu de données moléculaires représente actuellement ~80 % des collections techniques franco-allemandes. Un prérequis pour tester le modèle in situ sera de compléter le génotypage de la collection de référence et d’évaluer la pertinence des seuils génétiques proposés. L’évaluation du modèle en conditions réelles devrait faire l’objet d’un second projet.
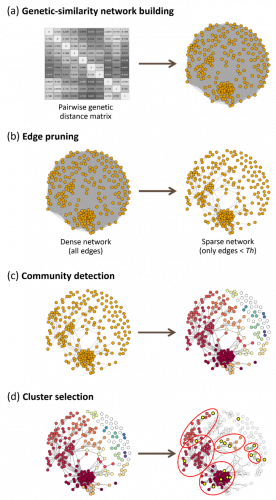
Figure 1. Description de l’approche en réseau. Une matrice de distances génétiques est calculée entre toutes les variétés puis transformée en réseau, dans lequel les nœuds (variétés) sont connectés par des liens dont le poids correspond à la distance génétique entre les nœuds (a). Tous les liens correspondant à une distance génétique supérieure à un seuil prédéfini (GDTh) sont supprimés pour simplifier le réseau (b) et un algorithme de détection de communauté est exécuté pour identifier des groupes de variétés apparentées (c). Seuls les groupes comprenant au moins une variété candidate (cercles jaunes) sont inclus dans les essais au champ (d).
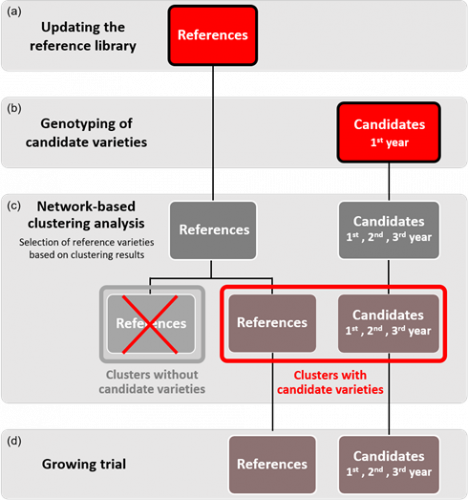
Figure 2. Vue schématique du modèle proposé pour l’intégration des données moléculaires dans l’examen DHS des variétés de colza d’hiver. Chaque année, les génotypes de la collection de référence sont mis à jour pour inclure les variétés récemment ajoutées (a). Une fois toutes les demandes reçues, les candidates sont génotypées (b) et une matrice de distances génétiques est calculée entre l’ensemble des variétés (candidates et les variétés de la collection de référence mise à jour). La matrice est ensuite analysée à l’aide de l’approche en réseau qui renvoie un tableau avec les résultats de regroupement (c). Seules les groupes comprenant une ou plusieurs variétés candidates sont sélectionnés pour être inclus dans les essais en culture (d).
Projet co-financé par l’OCVV et coordonné par le GEVES, en collaboration avec le Bundessortenamt.